KMT2A
| KMT2A | |||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||
| |||||||||||||||||
| Ідентифікатори | |||||||||||||||||
| Символи | KMT2A, ALL-1, CXXC7, HRX, HTRX1, MLL, MLL/GAS7, MLL1, MLL1A, TET1-MLL, TRX1, WDSTS, MLL-AF9, lysine methyltransferase 2A, Histone-lysine N-methyltransferase HRX, ALL1, HTRX | ||||||||||||||||
| Зовнішні ІД | MGI: 96995 HomoloGene: 4338 GeneCards: KMT2A | ||||||||||||||||
| Пов'язані генетичні захворювання | |||||||||||||||||
| Wiedemann-Steiner syndrome[1] | |||||||||||||||||
| |||||||||||||||||
| Шаблон експресії | |||||||||||||||||
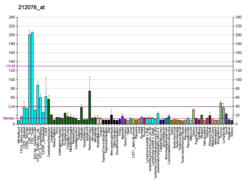   | |||||||||||||||||
| Більше даних | |||||||||||||||||
| Ортологи | |||||||||||||||||
| Види | Людина | Миша | |||||||||||||||
| Entrez |
|
| |||||||||||||||
| Ensembl |
|
| |||||||||||||||
| UniProt |
|
| |||||||||||||||
| RefSeq (мРНК) |
|
| |||||||||||||||
| RefSeq (білок) |
|
| |||||||||||||||
| Локус (UCSC) | Хр. 11: 118.44 – 118.53 Mb | Хр. 9: 44.71 – 44.79 Mb | |||||||||||||||
| PubMed search | [2] | [3] | |||||||||||||||
| Вікідані | |||||||||||||||||
| |||||||||||||||||
KMT2A (англ. Lysine methyltransferase 2A) – білок, який кодується однойменним геном, розташованим у людей на короткому плечі 11-ї хромосоми.[4] Довжина поліпептидного ланцюга білка становить 3 969 амінокислот, а молекулярна маса — 431 764[5].
| 10 | 20 | 30 | 40 | 50 | ||||
|---|---|---|---|---|---|---|---|---|
| MAHSCRWRFP | ARPGTTGGGG | GGGRRGLGGA | PRQRVPALLL | PPGPPVGGGG | ||||
| PGAPPSPPAV | AAAAAAAGSS | GAGVPGGAAA | ASAASSSSAS | SSSSSSSSAS | ||||
| SGPALLRVGP | GFDAALQVSA | AIGTNLRRFR | AVFGESGGGG | GSGEDEQFLG | ||||
| FGSDEEVRVR | SPTRSPSVKT | SPRKPRGRPR | SGSDRNSAIL | SDPSVFSPLN | ||||
| KSETKSGDKI | KKKDSKSIEK | KRGRPPTFPG | VKIKITHGKD | ISELPKGNKE | ||||
| DSLKKIKRTP | SATFQQATKI | KKLRAGKLSP | LKSKFKTGKL | QIGRKGVQIV | ||||
| RRRGRPPSTE | RIKTPSGLLI | NSELEKPQKV | RKDKEGTPPL | TKEDKTVVRQ | ||||
| SPRRIKPVRI | IPSSKRTDAT | IAKQLLQRAK | KGAQKKIEKE | AAQLQGRKVK | ||||
| TQVKNIRQFI | MPVVSAISSR | IIKTPRRFIE | DEDYDPPIKI | ARLESTPNSR | ||||
| FSAPSCGSSE | KSSAASQHSS | QMSSDSSRSS | SPSVDTSTDS | QASEEIQVLP | ||||
| EERSDTPEVH | PPLPISQSPE | NESNDRRSRR | YSVSERSFGS | RTTKKLSTLQ | ||||
| SAPQQQTSSS | PPPPLLTPPP | PLQPASSISD | HTPWLMPPTI | PLASPFLPAS | ||||
| TAPMQGKRKS | ILREPTFRWT | SLKHSRSEPQ | YFSSAKYAKE | GLIRKPIFDN | ||||
| FRPPPLTPED | VGFASGFSAS | GTAASARLFS | PLHSGTRFDM | HKRSPLLRAP | ||||
| RFTPSEAHSR | IFESVTLPSN | RTSAGTSSSG | VSNRKRKRKV | FSPIRSEPRS | ||||
| PSHSMRTRSG | RLSSSELSPL | TPPSSVSSSL | SISVSPLATS | ALNPTFTFPS | ||||
| HSLTQSGESA | EKNQRPRKQT | SAPAEPFSSS | SPTPLFPWFT | PGSQTERGRN | ||||
| KDKAPEELSK | DRDADKSVEK | DKSRERDRER | EKENKRESRK | EKRKKGSEIQ | ||||
| SSSALYPVGR | VSKEKVVGED | VATSSSAKKA | TGRKKSSSHD | SGTDITSVTL | ||||
| GDTTAVKTKI | LIKKGRGNLE | KTNLDLGPTA | PSLEKEKTLC | LSTPSSSTVK | ||||
| HSTSSIGSML | AQADKLPMTD | KRVASLLKKA | KAQLCKIEKS | KSLKQTDQPK | ||||
| AQGQESDSSE | TSVRGPRIKH | VCRRAAVALG | RKRAVFPDDM | PTLSALPWEE | ||||
| REKILSSMGN | DDKSSIAGSE | DAEPLAPPIK | PIKPVTRNKA | PQEPPVKKGR | ||||
| RSRRCGQCPG | CQVPEDCGVC | TNCLDKPKFG | GRNIKKQCCK | MRKCQNLQWM | ||||
| PSKAYLQKQA | KAVKKKEKKS | KTSEKKDSKE | SSVVKNVVDS | SQKPTPSARE | ||||
| DPAPKKSSSE | PPPRKPVEEK | SEEGNVSAPG | PESKQATTPA | SRKSSKQVSQ | ||||
| PALVIPPQPP | TTGPPRKEVP | KTTPSEPKKK | QPPPPESGPE | QSKQKKVAPR | ||||
| PSIPVKQKPK | EKEKPPPVNK | QENAGTLNIL | STLSNGNSSK | QKIPADGVHR | ||||
| IRVDFKEDCE | AENVWEMGGL | GILTSVPITP | RVVCFLCASS | GHVEFVYCQV | ||||
| CCEPFHKFCL | EENERPLEDQ | LENWCCRRCK | FCHVCGRQHQ | ATKQLLECNK | ||||
| CRNSYHPECL | GPNYPTKPTK | KKKVWICTKC | VRCKSCGSTT | PGKGWDAQWS | ||||
| HDFSLCHDCA | KLFAKGNFCP | LCDKCYDDDD | YESKMMQCGK | CDRWVHSKCE | ||||
| NLSDEMYEIL | SNLPESVAYT | CVNCTERHPA | EWRLALEKEL | QISLKQVLTA | ||||
| LLNSRTTSHL | LRYRQAAKPP | DLNPETEESI | PSRSSPEGPD | PPVLTEVSKQ | ||||
| DDQQPLDLEG | VKRKMDQGNY | TSVLEFSDDI | VKIIQAAINS | DGGQPEIKKA | ||||
| NSMVKSFFIR | QMERVFPWFS | VKKSRFWEPN | KVSSNSGMLP | NAVLPPSLDH | ||||
| NYAQWQEREE | NSHTEQPPLM | KKIIPAPKPK | GPGEPDSPTP | LHPPTPPILS | ||||
| TDRSREDSPE | LNPPPGIEDN | RQCALCLTYG | DDSANDAGRL | LYIGQNEWTH | ||||
| VNCALWSAEV | FEDDDGSLKN | VHMAVIRGKQ | LRCEFCQKPG | ATVGCCLTSC | ||||
| TSNYHFMCSR | AKNCVFLDDK | KVYCQRHRDL | IKGEVVPENG | FEVFRRVFVD | ||||
| FEGISLRRKF | LNGLEPENIH | MMIGSMTIDC | LGILNDLSDC | EDKLFPIGYQ | ||||
| CSRVYWSTTD | ARKRCVYTCK | IVECRPPVVE | PDINSTVEHD | ENRTIAHSPT | ||||
| SFTESSSKES | QNTAEIISPP | SPDRPPHSQT | SGSCYYHVIS | KVPRIRTPSY | ||||
| SPTQRSPGCR | PLPSAGSPTP | TTHEIVTVGD | PLLSSGLRSI | GSRRHSTSSL | ||||
| SPQRSKLRIM | SPMRTGNTYS | RNNVSSVSTT | GTATDLESSA | KVVDHVLGPL | ||||
| NSSTSLGQNT | STSSNLQRTV | VTVGNKNSHL | DGSSSSEMKQ | SSASDLVSKS | ||||
| SSLKGEKTKV | LSSKSSEGSA | HNVAYPGIPK | LAPQVHNTTS | RELNVSKIGS | ||||
| FAEPSSVSFS | SKEALSFPHL | HLRGQRNDRD | QHTDSTQSAN | SSPDEDTEVK | ||||
| TLKLSGMSNR | SSIINEHMGS | SSRDRRQKGK | KSCKETFKEK | HSSKSFLEPG | ||||
| QVTTGEEGNL | KPEFMDEVLT | PEYMGQRPCN | NVSSDKIGDK | GLSMPGVPKA | ||||
| PPMQVEGSAK | ELQAPRKRTV | KVTLTPLKME | NESQSKNALK | ESSPASPLQI | ||||
| ESTSPTEPIS | ASENPGDGPV | AQPSPNNTSC | QDSQSNNYQN | LPVQDRNLML | ||||
| PDGPKPQEDG | SFKRRYPRRS | ARARSNMFFG | LTPLYGVRSY | GEEDIPFYSS | ||||
| STGKKRGKRS | AEGQVDGADD | LSTSDEDDLY | YYNFTRTVIS | SGGEERLASH | ||||
| NLFREEEQCD | LPKISQLDGV | DDGTESDTSV | TATTRKSSQI | PKRNGKENGT | ||||
| ENLKIDRPED | AGEKEHVTKS | SVGHKNEPKM | DNCHSVSRVK | TQGQDSLEAQ | ||||
| LSSLESSRRV | HTSTPSDKNL | LDTYNTELLK | SDSDNNNSDD | CGNILPSDIM | ||||
| DFVLKNTPSM | QALGESPESS | SSELLNLGEG | LGLDSNREKD | MGLFEVFSQQ | ||||
| LPTTEPVDSS | VSSSISAEEQ | FELPLELPSD | LSVLTTRSPT | VPSQNPSRLA | ||||
| VISDSGEKRV | TITEKSVASS | ESDPALLSPG | VDPTPEGHMT | PDHFIQGHMD | ||||
| ADHISSPPCG | SVEQGHGNNQ | DLTRNSSTPG | LQVPVSPTVP | IQNQKYVPNS | ||||
| TDSPGPSQIS | NAAVQTTPPH | LKPATEKLIV | VNQNMQPLYV | LQTLPNGVTQ | ||||
| KIQLTSSVSS | TPSVMETNTS | VLGPMGGGLT | LTTGLNPSLP | TSQSLFPSAS | ||||
| KGLLPMSHHQ | HLHSFPAATQ | SSFPPNISNP | PSGLLIGVQP | PPDPQLLVSE | ||||
| SSQRTDLSTT | VATPSSGLKK | RPISRLQTRK | NKKLAPSSTP | SNIAPSDVVS | ||||
| NMTLINFTPS | QLPNHPSLLD | LGSLNTSSHR | TVPNIIKRSK | SSIMYFEPAP | ||||
| LLPQSVGGTA | ATAAGTSTIS | QDTSHLTSGS | VSGLASSSSV | LNVVSMQTTT | ||||
| TPTSSASVPG | HVTLTNPRLL | GTPDIGSISN | LLIKASQQSL | GIQDQPVALP | ||||
| PSSGMFPQLG | TSQTPSTAAI | TAASSICVLP | STQTTGITAA | SPSGEADEHY | ||||
| QLQHVNQLLA | SKTGIHSSQR | DLDSASGPQV | SNFTQTVDAP | NSMGLEQNKA | ||||
| LSSAVQASPT | SPGGSPSSPS | SGQRSASPSV | PGPTKPKPKT | KRFQLPLDKG | ||||
| NGKKHKVSHL | RTSSSEAHIP | DQETTSLTSG | TGTPGAEAEQ | QDTASVEQSS | ||||
| QKECGQPAGQ | VAVLPEVQVT | QNPANEQESA | EPKTVEEEES | NFSSPLMLWL | ||||
| QQEQKRKESI | TEKKPKKGLV | FEISSDDGFQ | ICAESIEDAW | KSLTDKVQEA | ||||
| RSNARLKQLS | FAGVNGLRML | GILHDAVVFL | IEQLSGAKHC | RNYKFRFHKP | ||||
| EEANEPPLNP | HGSARAEVHL | RKSAFDMFNF | LASKHRQPPE | YNPNDEEEEE | ||||
| VQLKSARRAT | SMDLPMPMRF | RHLKKTSKEA | VGVYRSPIHG | RGLFCKRNID | ||||
| AGEMVIEYAG | NVIRSIQTDK | REKYYDSKGI | GCYMFRIDDS | EVVDATMHGN | ||||
| AARFINHSCE | PNCYSRVINI | DGQKHIVIFA | MRKIYRGEEL | TYDYKFPIED | ||||
| ASNKLPCNCG | AKKCRKFLN |
Кодований геном білок за функціями належить до трансфераз, регуляторів хроматину, метилтрансфераз, фосфопротеїнів. Задіяний у таких біологічних процесах, як апоптоз, транскрипція, регуляція транскрипції, біологічні ритми, ацетилювання, альтернативний сплайсинг. Білок має сайт для зв'язування з іонами металів, іоном цинку, ДНК, S-аденозил-L-метіоніном. Локалізований у ядрі.
Література
- Tkachuk D.C., Kohler S., Cleary M.L. (1992). Involvement of a homolog of Drosophila trithorax by 11q23 chromosomal translocations in acute leukemias. Cell. 71: 691—700. PMID 1423624 DOI:10.1016/0092-8674(92)90602-9
- Hsieh J.J.-D., Ernst P., Erdjument-Bromage H., Tempst P., Korsmeyer S.J. (2003). Proteolytic cleavage of MLL generates a complex of N- and C-terminal fragments that confers protein stability and subnuclear localization. Mol. Cell. Biol. 23: 186—194. PMID 12482972 DOI:10.1128/MCB.23.1.186-194.2003
- Hsieh J.J.-D., Cheng E.H.-Y., Korsmeyer S.J. (2003). Taspase1: a threonine aspartase required for cleavage of MLL and proper HOX gene expression. Cell. 115: 293—303. PMID 14636557 DOI:10.1016/S0092-8674(03)00816-X
- Patel A., Dharmarajan V., Vought V.E., Cosgrove M.S. (2009). On the mechanism of multiple lysine methylation by the human mixed lineage leukemia protein-1 (MLL1) core complex. J. Biol. Chem. 284: 24242—24256. PMID 19556245 DOI:10.1074/jbc.M109.014498
- De Guzman R.N., Goto N.K., Dyson H.J., Wright P.E. (2006). Structural basis for cooperative transcription factor binding to the CBP coactivator. J. Mol. Biol. 355: 1005—1013. PMID 16253272 DOI:10.1016/j.jmb.2005.09.059
- Patel A., Dharmarajan V., Cosgrove M.S. (2008). Structure of WDR5 bound to mixed lineage leukemia protein-1 peptide. J. Biol. Chem. 283: 32158—32161. PMID 18829459 DOI:10.1074/jbc.C800164200
Примітки
- ↑ Захворювання, генетично пов'язані з KMT2A переглянути/редагувати посилання на ВікіДаних.
- ↑ Human PubMed Reference:.
- ↑ Mouse PubMed Reference:.
- ↑ HUGO Gene Nomenclature Commitee, HGNC:7132 (англ.) . Процитовано 8 вересня 2017.
- ↑ UniProt, Q03164 (англ.) . Архів оригіналу за 4 вересня 2017. Процитовано 8 вересня 2017.
Див. також
- Хромосома 11
 | Це незавершена стаття про білки. Ви можете допомогти проєкту, виправивши або дописавши її. |
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||










